Francisella novicida由来Cpf1によるイネおよびタバコゲノムの高効率な標的変異導入
Efficient targeted mutagenesis of rice and tobacco genomes using Cpf1 from Francisella novicida
2016年12月1日 Scientific Reports 6 : 38169 doi: 10.1038/srep38169
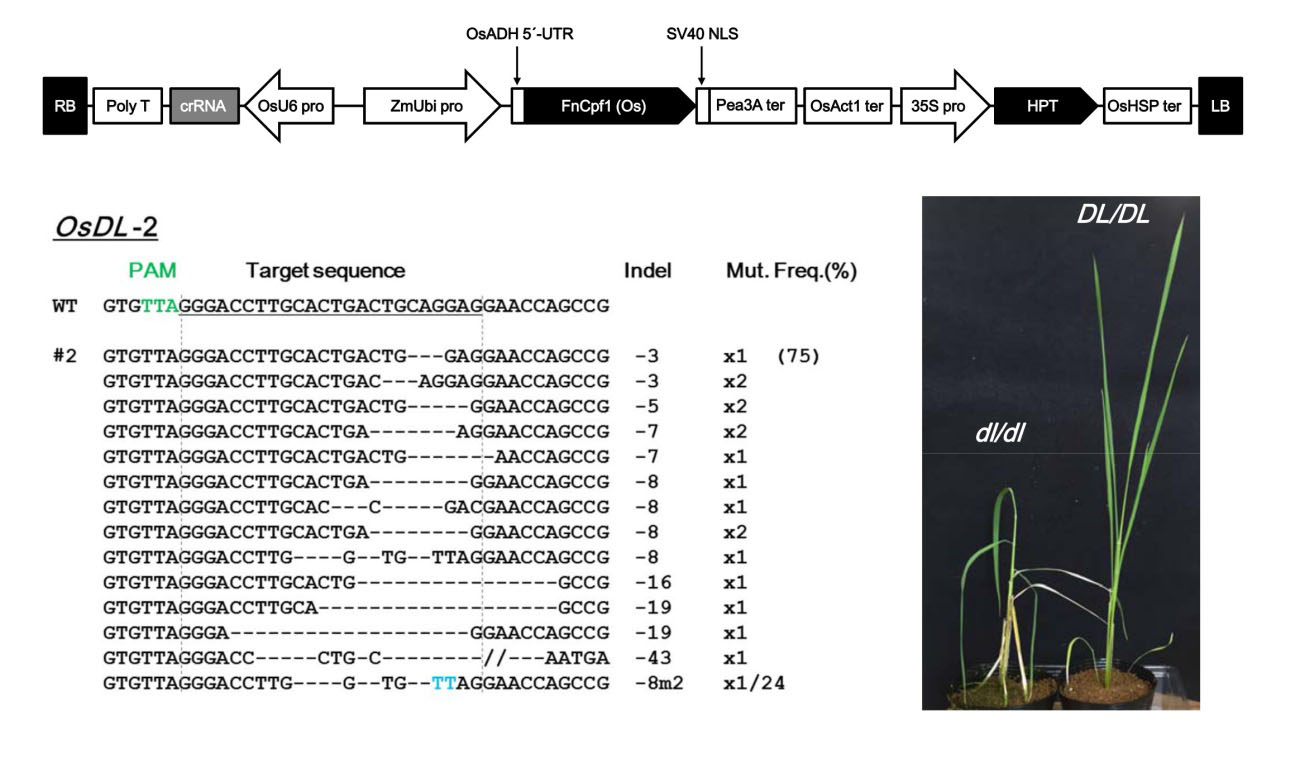
現在、植物を含むさまざまな生物のゲノム編集を行うために、CRISPR/Cas9システムが幅広く利用されている。Cpf1(CRISPR from Prevotella and Francisella 1)は、新しく特性が明らかにされたRNA誘導型エンドヌクレアーゼであり、Cas9とは異なる二つの特徴を持つ。第一に、Cas9はグアニジンに富むPAM(protospacer adjacent motif)を認識するが、Cpf1はチミジンに富むPAMを認識することから、Cpf1は、Cas9が不得意であったATリッチなゲノム領域を標的とする配列特異的ヌクレアーゼとして利用可能と考えられる。第二に、Cas9によるDNA切断で生じるのは平滑末端であるが、Cpf1は5'-突出のDNA末端を生じる。DNAの「粘着」末端は、相補的なDNA末端の利用によってCpf1切断部位への目的DNA断片の挿入効率を向上させることが期待される。従って、Cpf1は精密なゲノム編集の強力なツールとなる可能性を持つ。Cpf1が植物ゲノムの編集に応用可能かどうかを評価するため、我々は、既知のCpf1タンパク質のなかでより短いPAM(TTN)を認識するFrancisella novicida由来のCpf1を選出し(FnCpf1)、それをタバコおよびイネの選択的な変異導入に用いた。その結果、FnCpf1とともにcrRNAを発現する形質転換植物で標的変異導入が生じたことが明らかになった。観察された変異としては、標的領域の欠失が最も高頻度であった。今回の結果は、FnCpf1が植物のゲノム編集に広く応用可能であることを示している。
Corresponding Author
CRISPR/Cas9 systems are nowadays applied extensively to effect genome editing in various organisms including plants. CRISPR from Prevotella and Francisella 1 (Cpf1) is a newly characterized RNA-guided endonuclease that has two distinct features as compared to Cas9. First, Cpf1 utilizes a thymidine-rich protospacer adjacent motif (PAM) while Cas9 prefers a guanidine-rich PAM. Cpf1 could be used as a sequence-specific nuclease to target AT-rich regions of a genome that Cas9 had difficulty accessing. Second, Cpf1 generates DNA ends with a 5′ overhang, whereas Cas9 creates blunt DNA ends after cleavage. “Sticky” DNA ends should increase the efficiency of insertion of a desired DNA fragment into the Cpf1-cleaved site using complementary DNA ends. Therefore, Cpf1 could be a potent tool for precise genome engineering. To evaluate whether Cpf1 can be applied to plant genome editing, we selected Cpf1 from Francisella novicida (FnCpf1), which recognizes a shorter PAM (TTN) within known Cpf1 proteins, and applied it to targeted mutagenesis in tobacco and rice. Our results show that targeted mutagenesis had occurred in transgenic plants expressing FnCpf1 with crRNA. Deletions of the targeted region were the most frequently observed mutations. Our results demonstrate that FnCpf1 can be applied successfully to genome engineering in plants.

