リカレンスプロットを用いた1細胞染色体構造の3次元的な再構成
Three-dimensional reconstruction of single-cell chromosome structure using recurrence plots
2016年10月11日 Scientific Reports 6 : 34982 doi: 10.1038/srep34982
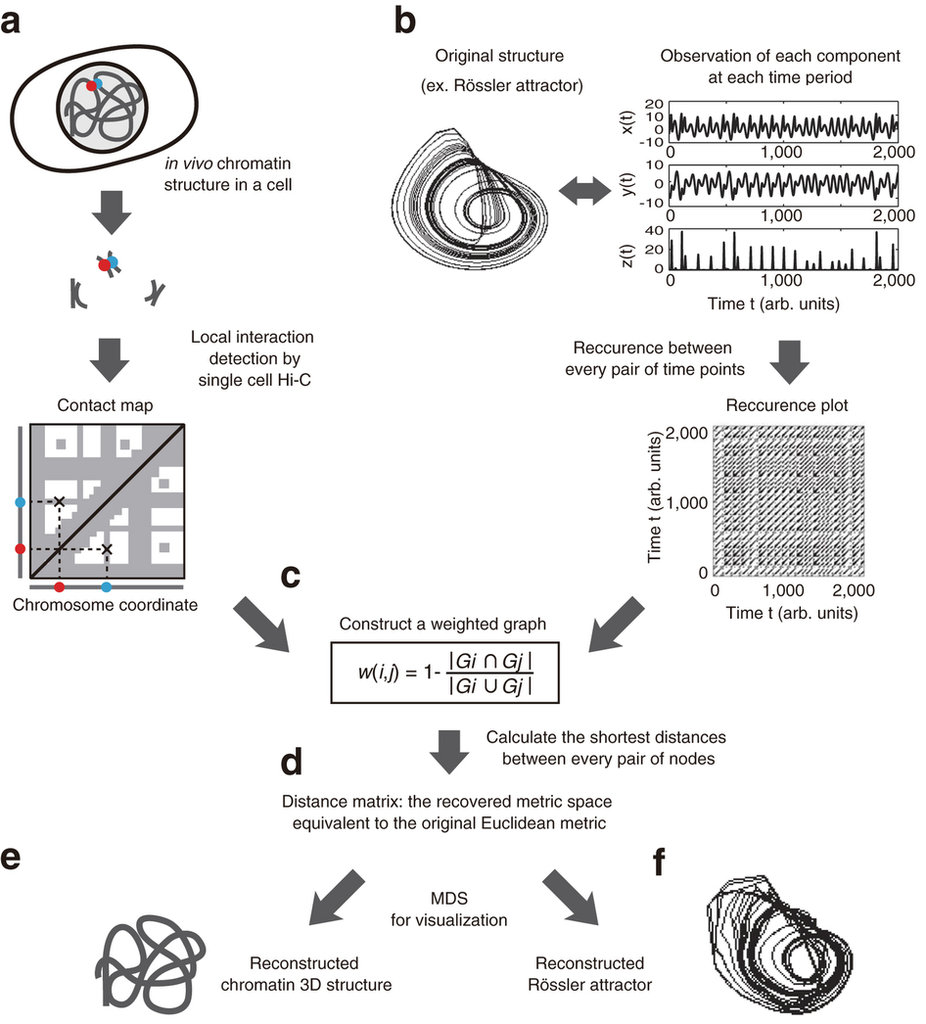
個々の細胞において染色体DNAの3次元構造が異なることが知られており、細胞間の個性をもたらす一要因となっている。今回、1細胞におけるゲノムDNAの3次元構造を再構成する新しい数学的手法を開発した。この手法(RPR法)は、ゲノムワイドなchromosome conformation capture(Hi-C)データから得られる染色体の部分同士の隣接情報に対して、非線形時系列解析のための数学的手法であるリカレンスプロットを適用するものである。この手法を用いることで、データ欠損の激しい1細胞由来のHi-C情報からでも、細胞固有の構造を迅速かつ一義的に再構成できるというメリットが得られる。
Corresponding Authors
Single-cell analysis of the three-dimensional (3D) chromosome structure can reveal cell-to-cell variability in genome activities. Here, we propose to apply recurrence plots, a mathematical method of nonlinear time series analysis, to reconstruct the 3D chromosome structure of a single cell based on information of chromosomal contacts from genome-wide chromosome conformation capture (Hi-C) data. This recurrence plot-based reconstruction (RPR) method enables rapid reconstruction of a unique structure in single cells, even from incomplete Hi-C information.

