総合的分子相互作用地図に基づくネットワーク分析により酵母のストレス応答経路上のロバスト制御構造を解明する
Network analyses based on comprehensive molecular interaction maps reveal robust control structures in yeast stress response pathways
2016年1月7日 npj Systems Biology and Applications 2, Article number:15018 (2016) doi:10.1038/npjsba.2015.18
コンピュータモデリング:分子機構を地図化する
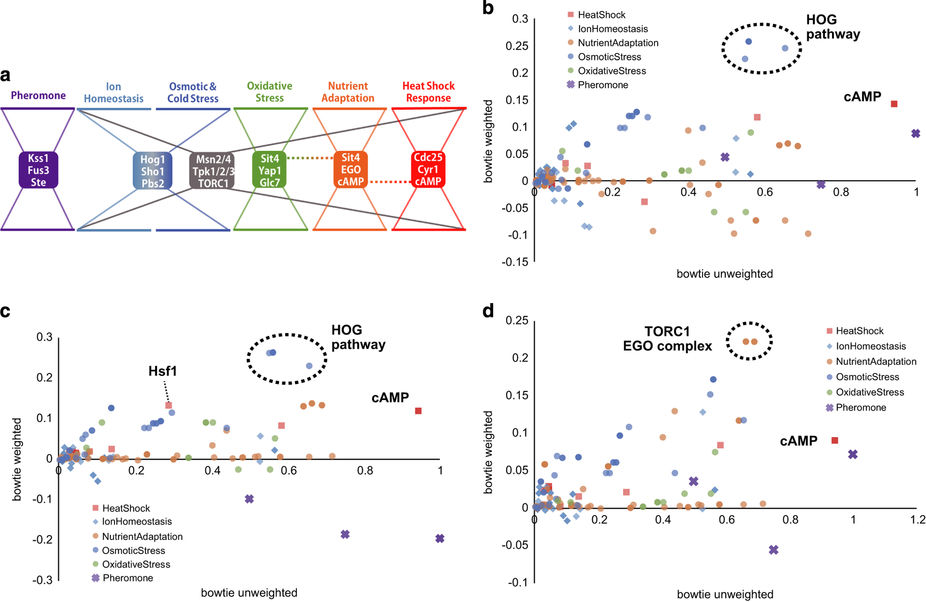
精選された実験データから組み立てたコンピュータモデルにより、酵母細胞がストレッサーに反応する機構についての理解が得られる。生物学的プロセスはいずれも、数々の異なる分子的因子間の複雑な相互作用によってもたらされており、こうした相互作用をモデル化することは非常に難しい仕事になりかねない。日本のシステム・バイオロジー研究機構とインドのPersistent Systems Limited社による日印合同チームは、本研究において、公表されている論文やデータの大規模なキュレーションにより、高度な生物学的ネットワークを再構成できる方法を明らかにしている。同チームの研究者たちは酵母が熱や栄養失調などのストレッサーへの適応に使用している経路に注目し、発表されている研究900件以上の調査結果を機械読み取り可能なビジュアルフォーマットに変換した。この知識ベースをネットワーク分析することにより、ストレス応答ネットワークの活動を制御する主要因とそうした制御が発生するメカニズムを解明した。
Computer modeling: Putting molecular mechanisms on the map
Computer models assembled from curated experimental data offer insights into the mechanisms by which yeast cells respond to stressors. Every biological process is dependent on complex interactions between a host of different molecular factors, and modeling this interplay can pose a daunting challenge. A Japan-India team formed by Japan’s Systems Biology Institute and Persistent Systems Limited in India has now demonstrated how sophisticated biological networks can be reconstructed from a large-scale curation of publicly available articles and data. The researchers focused on the pathways that yeast use to adapt to stressors such as heat and nutrient deprivation, translating findings from more than 900 previously published studies into a machine-readable, visual format. Network analysis on this knowledge base revealed key factors that regulate the activity of stress-response networks and the mechanisms by which this regulation occurs.

