サメのゲノムが挑む、進化と自然の謎
工樂 樹洋
2018年11月号掲載
軟骨魚類板鰓(ばんさい)類のサメ類は、その名の通り、硬い骨を持たない。ヒトの属する硬骨脊椎動物の祖先とは、4億5000万年前に分岐した。日本近海には、映画で有名なホホジロザメ、巨大なジンベエザメ、ダイバーに人気のシュモクザメをはじめ、約150種が生息している。このほど、理化学研究所生命機能科学研究センター分子配列比較解析ユニットの工樂樹洋ユニットリーダーらを中心とする、沖縄美ら海水族館、海遊館、大阪市立大学、東京大学の共同研究チームが、ジンベエザメ、トラザメ、イヌザメの全ゲノムを解析し、脊椎動物の進化やサメの生態についてさまざまな知見を得た。成果は、Nature Ecology & Evolution 11月号に発表された。
―― なぜ、サメを対象にしたのでしょう?
工樂氏: 脊椎動物のメジャーなグループのほとんどはどれかの種で全ゲノム解析が行われていますが、サメ類ではまとまった解析が皆無でした。しかし、脊椎動物の祖先の姿を知りたければ、サメ類を避けて通ることはできません。というのもサメ類は、脊椎動物共通の祖先から早い時期に分岐している、つまり我々哺乳類の祖先と別れてかなり時間が経っているからです(図1)。サメ類より前に分岐した生物に、ヤツメウナギなどの円口類がいます(図1)。私は国際コンソーシアムの中心メンバーの1 人として2013年にヤツメウナギのゲノム解析の論文を発表しましたが1、円口類のゲノムは、進化距離以上に遺伝子の数や配列が変化していることが多く、やっかいです。一方、サメ類は、情報が少ない時代から分子進化速度が遅く祖先からの遺伝子の配列やレパートリの変化が少ないといわれていました。このため、脊椎動物の祖先をたどるには、格好のターゲットとなります。

サメ類はエイ類とともに軟骨魚類のうち板鰓類というグループに属する。いっぽう、以前からゲノム情報が公表されていたゾウギンザメはギンザメ類(全頭類)に属する。 | 拡大する
それに、学生の頃からサメに興味を持っており、いつかはサメのゲノムを調べたいと思っていました。なのに、何年経っても、まとまったゲノム情報が出てきませんでした。自分で手掛けることになるとは予想しませんでしたが、自分たちで次世代シーケンサーを使うノウハウが蓄積するなど、実験技術やデータ解析スキルなどのすべての条件が整ったことで満を持して着手し、一通りの解析を行うことができました。
―― ゲノムサイズが大きいということですが。
工樂氏: 全てのサメで大きいわけではありません。今回、トラザメが6.67ギガ塩基対、イヌザメが4.73ギガ塩基対と推定しましたが、大きめであることは以前から知られていました。ジンベエザメは我々が初めて調べたのですが、3.75ギガ塩基対です。ただ、サメ類のゲノムが大きいのは、ゲノム倍加(脊椎動物の進化過程では全ゲノムが倍加する現象が何回か起きている)によるものではありません。遺伝子をコードしていない、ノンコーディング領域が増えているのです(図2)。
―― ノンコーディング領域が増えた理由は?
1番の謎です。サメの独特の生息環境が関係しているのかもしれません。これは推測ですが、天敵がいない深海での暮らしでは、外界に反応して素早く遺伝子を働かせる必要がなくなり、転写やスプライシング、そして翻訳に時間がかかるゲノムの変化でも許容されたのかもしれません。「スローライフ」がゲノムの構造に表れているのかな、と。これは、さらに多様な環境に生息する種での解析などで今後確認したいと思っています。
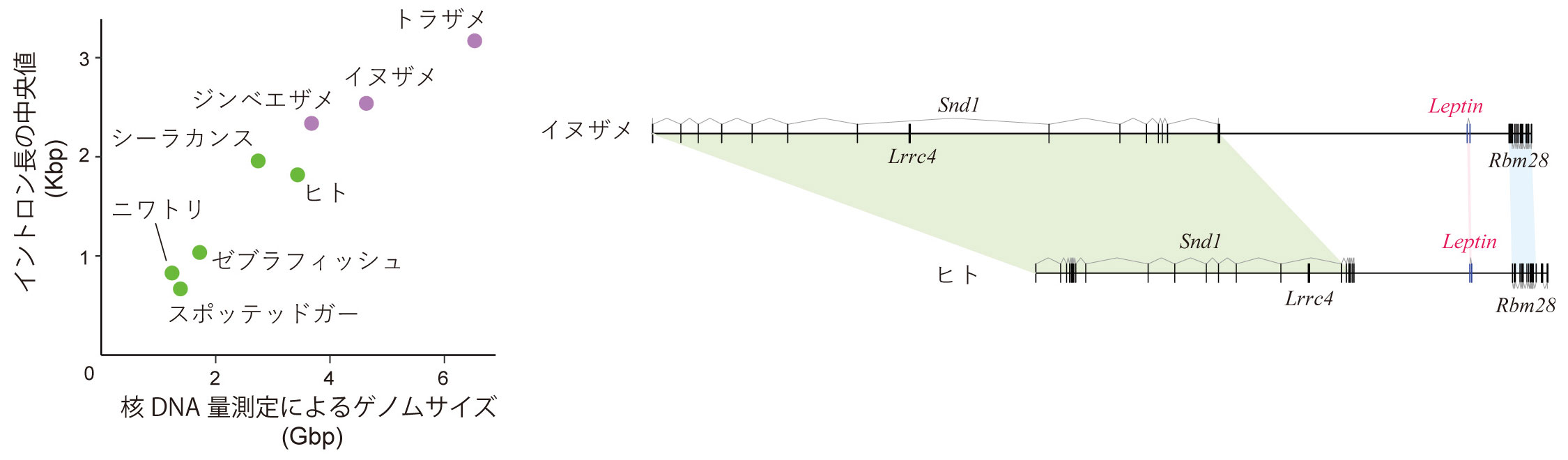
ゲノムサイズとイントロン長のおおよその相関は、今回解析したサメ3種を含めても成り立つ(左)。この図などから、サメにおけるゲノムサイズの肥大化は、サメ独自の系統での全ゲノム重複ではなく、ゲノム全体の弛緩によるものであることが示唆された。レプチン遺伝子の両端の遺伝子間距離やイントロンの長さにも、この傾向が表れている(右)。縦線がエキソンを示し、その間の領域がイントロン。 | 拡大する
―― 今回の解析は、いろいろな分野にわたっていますね。
工樂氏: 意図して、さまざまな話題を散りばめました。解析に当たっては、2014年に発表されたゾウギンザメのゲノム解析の論文2で硬骨の起源や免疫系が調べられていたので、これらはあえて避けました。サメ類のゲノム情報を手にしたからこそ初めて言えることを突き詰めようと、いろいろな分野から解析を進めました。
―― まずは、HoxC遺伝子群※がサメにあることを見つけましたね。
工樂氏: ゲノム情報がない時代に、サメ類はHoxCクラスターをまるごと持たないというのが定説になっていました。それが真実かを見極める必要を感じていたのです。ただ、HoxクラスターのA、B、Dは反復配列が少ないためにアセンブルするとゲノム配列が非常に繋がりやすいのですが、サメのHoxCは反復配列が多くしかもGC含量が多いため、シーケンスしづらく繋がりにくい。技術的には厄介ですが、進化学的には、ゲノム配列の読み取りが難しい部分ほど面白いと思っています。
今回の結果から、サメの種類によって異なるHoxC遺伝子のレパートリを持つことが示唆されました。もしかすると、サメの種間の形態の違いを説明するものかもしれません。
―― 内分泌系のホルモンについてはどうでしょう。
工樂氏: 際立った脊椎動物らしさの1つとして注目し、成長ホルモン、オキシトシン、プロラクチン、オレキシンなどの遺伝子セットを調べました。ヒトの肥満とのかかわりで脚光を浴びたレプチンについては、初めて軟骨魚類において遺伝子として働いていることを発見しました。配列上の類似性は高くないのですが、ゲノム上の隣の遺伝子がオーソログであることから(図2)、まさしくレプチンのオーソログであると結論づけました。サメには明確な脂肪細胞がなく、どの組織で活発に働いているのかはまだ分かっていません。
こうしたホルモンがサメ類で見つかり、およそ4億5千万年前のサメ類と我々の共通祖先ですでに内分泌系の多くの遺伝子が揃っていたことが分かったわけです。
―― 光受容体オプシンも調べていますね。
工樂氏: サメにはオプシンが少なそうだとはいわれていましたが、ゲノム全体をみて確かめる必要がありました。想定外だったのは、トラザメにロドプシン(光の明暗のみ感受)しか見つからなかったことです。これはサメ以外でもほとんど例がありません。また、ジンベエザメとイヌザメは、ロドプシンと赤色光を感受するオプシンだけを持っていました。しかもジンベエザメのロドプシンは、深海にも届く480nmの波長の光をキャッチできるように変化していました。このようなロドプシンと赤色光を感受するオプシンとを保持していることは、海面近くで餌を食べながら時折深い所に潜るという暮らし方に合致しています。このジンベエザメの暮らし方は最近になってやっと分かってきたところで、いまだ繁殖の記録が全くないなど、謎だらけの巨大生物です。
―― 一方、サメ類にはあって哺乳類にはない遺伝子があるということですが。
工樂氏:: はい。脊椎動物の祖先が持っており、サメのゲノムにも含まれるにもかかわらず、ヒトに至る進化のどこかの時点で失われた遺伝子が、300個以上あることが分かりました。何かが失われるその前の状態を映してくれる、これがサメの魅力の1つです。前述のように、サメ類は進化速度が遅くゲノムの二次的な変更が少ないので、遺伝子セットも脊椎動物の祖先のものに近いと考えられます。これまで、遺伝子の増え方は調べられてきましたが、減っていった遺伝子を調べた研究は限られています。
―― そのうちの1つがFoxG遺伝子グループということですね。
工樂氏:: FoxG1は、大脳新皮質の層構造を作るマスター遺伝子で、終脳の発生・分化に必要なことが分かっています。が、それ以外は、各生物に遺伝子が何個あるかすら整理されていませんでした。我々は初めて詳細な分子系統解析を行い、今回調べたサメ類とスポッテッドガー、シーラカンスはFoxG1、FoxG2、FoxG3を保持しているが、は虫類はFoxG1とFoxG2、哺乳類や鳥類はFoxG1しかないことを示しました(図3)。つまり、軟骨魚類の祖先が分岐した時点では、脊椎動物の祖先は3つのFoxG遺伝子を持っていたということです。その3つとも持っていて、すぐに実験を行うことができる種が、まさにサメ類にしかいませんでした。
FoxG1は脳神経系を中心に広く発現している一方、FoxG3はいくつかの脳神経節と網膜、FoxG2はいくつかの脳神経節のみでしか発現しておらず、互いに包含関係にあることが分かりました(図3)。FoxG1の広範囲での発現はこの遺伝子をどの種も保持しているという進化上の広がりにも対応し、アミノ酸配列も非常に保存的です。ところが、FoxG2とFoxG3は働く部位が狭く、一部の種ではゲノムから欠落している。そして、種間でアミノ酸配列が変わりやすいのです。ここまではよくあるパターンで想定内でした。
今回、新しく分かったのは、遺伝子の残りやすさがタンパク質コード領域以外の配列にも関係しているということです。トラザメとイヌザメ(進化的にヒトとマウスよりちょっと離れている)を比較すると、FoxG1は遺伝子の上流と下流のノンコーディング領域もある程度保存されていました(図3)。が、FoxG2、FoxG3は、そういった部分での類似性は限られていました。似た傾向は、反復配列の頻度やGC含量、そして、コーディング領域のアミノ酸配列に影響を与えない塩基座位(同義座位)の変化のスピードにも表れていました。ゲノムワイドな解析でもこの傾向を示し、我々は「Field disparity(ゲノムの場の不均一性)」仮説として別の論文3で報告しました。これまで、遺伝子の機能面での必要度が進化の過程での遺伝子の残りやすさを決めるとされてきましたが、それならば、機能に影響しない部分に差が出ることはありません。遺伝子の機能以外に、遺伝子が乗っているゲノム領域の性質によっても遺伝子の失われやすさが左右されると考えています。
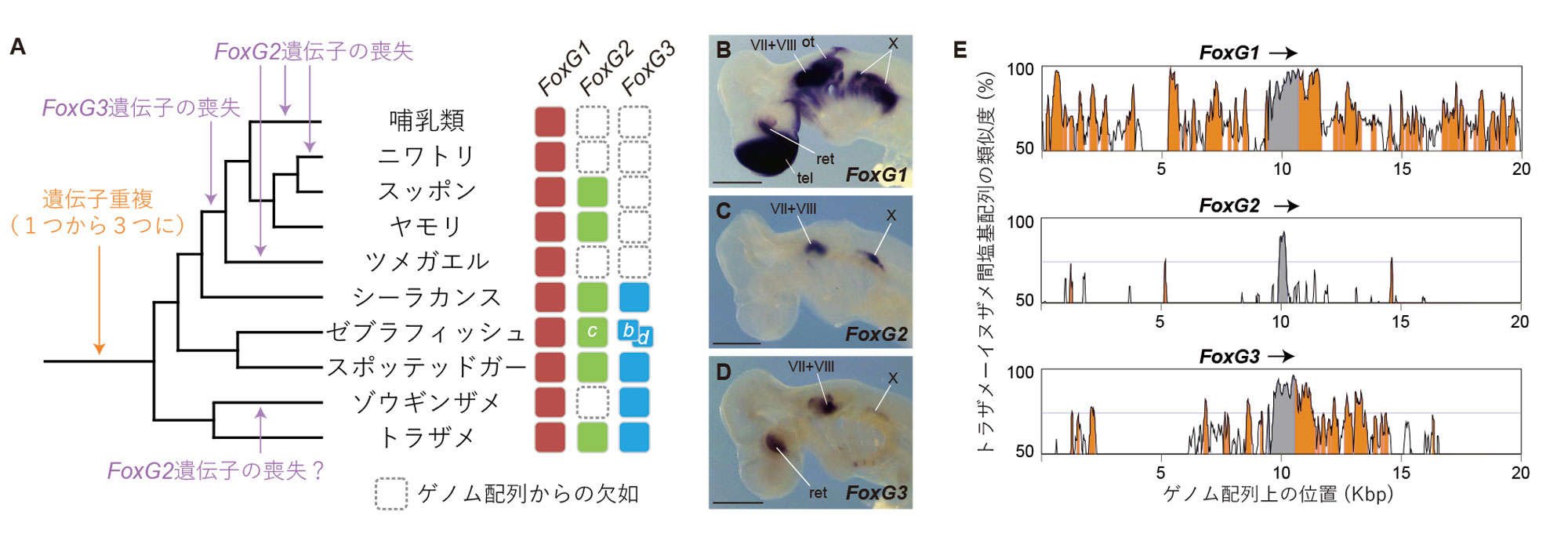
A. 遺伝子レパートリとその成立過程。B-D. トラザメ胚での発現パターン。VII+VIII:聴覚顔面神経節複合体、Ot:耳胞、X:迷走神経節、ret:網膜、tel:終脳。E. ゲノム配列の保存性。FoxG遺伝子のコーディング領域を矢印で表した。70%以上の塩基配列の類似性が見られるノンコーディング領域をオレンジで表した。FoxG遺伝子のコーディング領域は1つのエキソンに含まれることに注意。 | 拡大する
―― 今回の研究の意義は何でしょう?
工樂氏:: 今後、多くの人が利用して、いろいろな分子の研究が進むことです。これこそ、ゲノム解析の1番の価値です。
さらに、いずれこの研究が、水族館での計画的な飼育展示や自然界での生物の保全に役立つことも期待しています。そういう意味で、Nature Ecology & Evolutionを選択したことは、正解でした。自然界で起きていることへの興味や、人間はそれとどう付き合っていけばよいのかという懸念も念頭に置いて研究を進めてきたことの表明のようなものです。ゲノミクスや発生のジャーナルではここまでのフィット感は得られなかったと思います。実際、水産学の研究者や水族館関係者、そして保全に取り組む海外の研究者も、この論文に興味を持ってくれています。それに、シーラカンスのゲノムもガーのゲノムもインパクトの高いジャーナルに掲載されているので、我々もそういったジャーナルに出さないとサメに悪いなと思っていました(笑)。
―― お話を聞いてサメの面白さが分かりました。将来は、サメについてもっと解析したいのでは?
工樂氏:: ええ、やりたいですね。でも、今回の論文では、自らの経験や興味に固執せずいろいろな研究をしてきたからこそ、難しいゲノムを相手にしてさまざまな問いに切り込むことができました。そういう積み重ねひとつひとつが、結局はやりたいことに生きてくると思っています。今後もバランスをとりながら続けていくことになるでしょう。
―― ところで、小さい頃から、研究に興味を持っていたのですか?
工樂氏:: 科学者になりたいと思ったのは、小学生の頃です。文集に書いていました。遺伝子の研究に興味を持ったのは、高校3年の時、最初の師匠となる宮田隆さんの本、『分子進化学への招待(現在のタイトルは「分子からみた生物進化」)』を読んだことがきっかけです。多様な生き物に触れながら、縦横無尽にDNA情報を読み取ることができる今の環境は、理想に近いですね。
―― 最後に研究者を目指す若い人たちにメッセージを。
工樂氏: まずは、自分でキャリアを選べるように、技能を身につけることが肝要です。そしてその選択肢を自分で狭めないことです。もちろん、根底に自分がやりたいことがあっての話です。今でなくても、いつかは実現しようと。ほっておいても自分はいずれやる、自分がやらなくてはということにどれだけ邁進(まいしん)できるかです。それを大事にしてほしい。
それから、実験動物を用いた研究は、これまでの積み重ねが大事でこれからも続けられるべきではありますが、そこにとらわれず、自然界の謎にも直接目を向けてほしいです。今は、いろいろな手段で自然にアクセスできるようになり、新しい視野で研究できるチャンスが広がっています。Nature Ecology & Evolution の論文を読むと、フィールド系の研究が多いことが分かります。日本では、自然を舞台にした分子レベルの研究が少ないように思います。今やっと調べられるようになったことがたくさんありますので、新しい世代の研究者が挑むべき問いは尽きません。
―― ありがとうございました。
*HoxC遺伝子群
Hox遺伝子群は、動物の胚発生初期において、体の前後軸に沿った構造のパターン形成に関わる転写因子のクラスター。多くの場合、Hox1〜Hox13が番号順に並び、この番号順に頭部から尾部で発現する。つまり、Hox1は頭部で、Hox13 は尾部で、という具合に発現し、発現部位の形態形成に関わっている。それぞれの遺伝子は、ホメオボックスと呼ばれるDNA結合ドメインをコードする配列を持つ。脊椎動物では、進化の過程で2回の遺伝子重複により、HoxA、HoxB、HoxC、HoxDという4つのクラスターが存在する。
参考文献
- Smith, J. J. et al. Sequencing of the sea lamprey ( Petromyzon marinus) genome provides insights into vertebrate evolution. Nature Genetics 45, 415–421 (2013).
- Venkatesh, B. et al. Elephant shark genome provides unique insights into gnathostome evolution. Nature 505, 174–179 (2014).
- Hara, Y. et al. Madagascar ground gecko genome analysis characterizes asymmetric fates of duplicated genes. BMC Biology 16, 40 (2018).
インタビューを終えて
居室の棚にはジンベエザメの置物が飾られ、研究室の水槽にはかわいらしいイヌザメの子どもが泳いでいる。——ほんとうにサメが好きなのだと感じました。実際、お話を聞いていると、サメには謎が多く、私自身も面白いなと感じました。研究室のメンバーは別にずっとサメを追いかけてきたわけでもないが、こうした研究を通じて広範な研究を支える技術部門のスキルが向上すれば、という工樂先生。今後、サメをはじめ、自然界のさまざまな謎に迫ってほしいと思います。
聞き手は、田中明美(サイエンスライター)。
Nature Ecology & Evolution 掲載論文
Article: サメ類のゲノム解析がもたらした板鰓類の進化と脊椎動物の起源に関する手掛かり OPEN
Shark genomes provide insights into elasmobranch evolution and the origin of vertebrates
Nature Ecology & Evolution 2 : 1761–1771 doi:10.1038/s41559-018-0673-5 | Published online 08 October 2018
Author Profile
工樂 樹洋(くらく しげひろ)
理化学研究所生命機能科学研究センター分子配列比較解析ユニット ユニットリーダー
| 1999年 | 京都大学理学部卒業 |
| 2004年 | 京都大学大学院理学研究科博士課程単位取得認定退学(のち2005年博士(理学)) |
| 2005年 | 理化学研究所発生再生科学総合研究センター 研究員 |
| 2007年 | ドイツ・コンスタンツ大学 教員(Akademischer Rat) |
| 2012年 | 理化学研究所発生再生科学総合研究センター ユニットリーダー |
| 組織再編による所属名変更を経て、2018年より現職。 |
研究のモットーは、「流行りものに惑わされず、誰もやらない重要な研究を」。



