CRISPR-CasによるssODNを介した受精卵における大きなゲノム領域のノックイン
ssODN-mediated knock-in with CRISPR-Cas for large genomic regions in zygotes
2016年1月20日 Nature Communications 7 : 10431 doi: 10.1038/ncomms10431
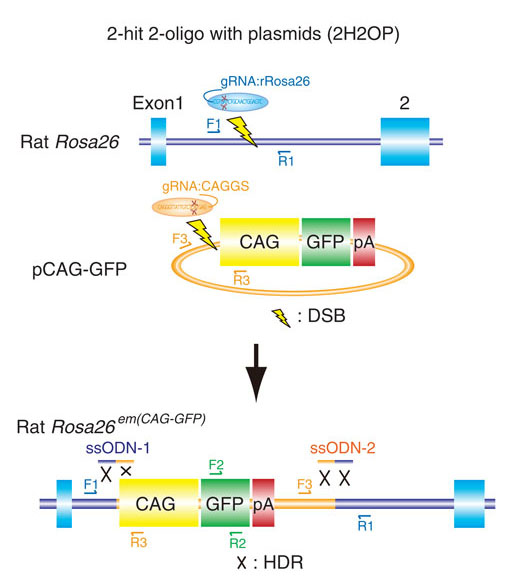
CRISPR-Casシステムは、遺伝子組換え動物の作出のための強力なツールである。しかし、相同組換えにより特定のゲノム部位にノックイン(KI)する方法は、受精卵では依然として困難である。本論文では、1本鎖オリゴデオキシヌクレオチド(ssODN)を用いたCRISPR-Casシステムにより、ラットにおける効率的な遺伝子KIを紹介している。一つ目は、1 kbのssODNを、ガイドRNA(gRNA)とCas9メッセンジャーRNAとともに注入し、ラットのThy1座位にGFP-KIを作り出した。二つ目は、2つのgRNAを2つの80bp ssODNを同時に用いて、ssODNによる末端結合を利用することで、5.5 kbの CAG-GFPベクターをRosa26座位に効率的に挿入した。また、このプロトコールによって、ヒトのSIRPA座位を含む200 kb BACをKIし、同時にラットのSirpa遺伝子をノックアウトすることができた。最後に、3つのgRNAおよび2つのssODNを用いて、58 kbのラットのCyp2d クラスターを 6.2 kbのヒトCYP2D6遺伝子に置き換えた。上記のssODNを用いたKIプロトコールは、相同組換え用アームを構築する必要がなく、あらゆる標的ゲノム部位にあらゆるドナーベクターを利用することが可能であるため、生体におけるゲノム編集操作をより平易にする。
Kazuto Yoshimi, Yayoi Kunihiro, Takehito Kaneko, Hitoshi Nagahora, Birger Voigt & Tomoji Mashimo

