Research Abstract
ヒト細胞におけるモチーフ標的定量プロテオミクスによるリン酸化修飾絶対化学量論の大規模解析
Large-scale determination of absolute phosphorylation stoichiometries in human cells by motif-targeting quantitative proteomics
2015年3月27日 Nature Communications 6 : 6622 doi: 10.1038/ncomms7622
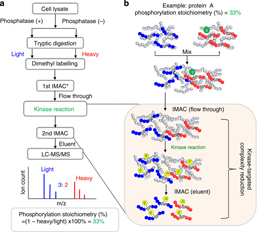
シグナル伝達ネットワーク動態をモデル化するためには、正確な大規模タンパク質リン酸化修飾定量法が必要である。今回我々は、リン酸化修飾の化学量論を測定するためのモチーフ標的定量法について述べる。プロテオーム規模のリン酸化修飾化学量論は、脱リン酸化と同位体によるタグ付け後に、酵素的にキナーゼ反応を行うという簡潔なリン酸化プロテオミクスのワークフローにより得ることができた。肺がん細胞におけるCK2、MAPKおよびEGFRを標的とする概念実証実験から、キナーゼ標的に基づく試料の複雑性低減という利点により、より詳細にリン酸化プロテオームを定量化できることが実証された。発現量の低いチロシンリン酸化部位366種を含む、1000を超えるリン酸化部位のリン酸化修飾化学量論を、高い再現性かつ少ない試料量で測定した。薬剤耐性と薬剤感受性の肺がん細胞の比較により、翻訳後修飾であるリン酸化の変化は、タンパク質レベルやメッセンジャーRNAレベルでの変化よりも非常に動的であることが明らかになり、薬剤耐性獲得に関連するキナーゼ–基質ネットワーク内に存在する薬剤標的候補が示唆された。
Chia-Feng Tsai, Yi-Ting Wang, Hsin-Yung Yen, Chih-Chiang Tsou, Wei-Chi Ku, Pei-Yi Lin, Hsuan-Yu Chen, Alexey I. Nesvizhskii, Yasushi Ishihama & Yu-Ju Chen
Corresponding Authors
Our ability to model the dynamics of signal transduction networks will depend on accurate methods to quantify levels of protein phosphorylation on a global scale. Here we describe a motif-targeting quantitation method for phosphorylation stoichiometry typing. Proteome-wide phosphorylation stoichiometry can be obtained by a simple phosphoproteomic workflow integrating dephosphorylation and isotope tagging with enzymatic kinase reaction. Proof-of-concept experiments using CK2-, MAPK- and EGFR-targeting assays in lung cancer cells demonstrate the advantage of kinase-targeted complexity reduction, resulting in deeper phosphoproteome quantification. We measure the phosphorylation stoichiometry of >1,000 phosphorylation sites including 366 low-abundance tyrosine phosphorylation sites, with high reproducibility and using small sample sizes. Comparing drug-resistant and sensitive lung cancer cells, we reveal that post-translational phosphorylation changes are significantly more dramatic than those at the protein and messenger RNA levels, and suggest potential drug targets within the kinase–substrate network associated with acquired drug resistance.

